Forscher verwenden die Genome von 241 Arten, um den Baum des Lebens bei Säugetieren neu zu definieren
Die Forschung, die von einem Team von Wissenschaftlern des College of Veterinary Medicine and Biomedical Sciences der Texas A&M geleitet wurde, hat eine hitzige wissenschaftliche Debatte über die Geschichte der Diversifizierung von Säugetieren in Bezug auf das Aussterben der Nicht-Vogel-Dinosaurier angeheizt. Ihre Arbeit liefert eine definitive Antwort auf die evolutionäre Zeitachse der Säugetiere in den letzten 100 Millionen Jahren.
Die Studie veröffentlicht in WissenschaftenEs ist Teil einer Reihe von Artikeln, die von veröffentlicht werden Zoonomia-ProjektEin Konsortium von Wissenschaftlern aus der ganzen Welt verwendet den größten Datensatz von Säugetiergenomen in der Geschichte, um die Evolutionsgeschichte des menschlichen Genoms im Kontext der Evolutionsgeschichte von Säugetieren zu definieren. Ihr ultimatives Ziel ist es, die genetische Grundlage für Merkmale und Krankheiten bei Menschen und anderen Arten besser zu definieren.
Die Forschung der Texas A&M University unter der Leitung von Dr. William J. Murphy, Professor am Department of Veterinary Complementary Biosciences, und Dr. Nicole Foley, Associate Research Scientist in Murphys Labor – haben ihre Wurzeln in der Phylogenie, dem Zweig der Biologie, der sich mit den evolutionären Beziehungen und der Diversifizierung ausgestorbener Organismen befasst.
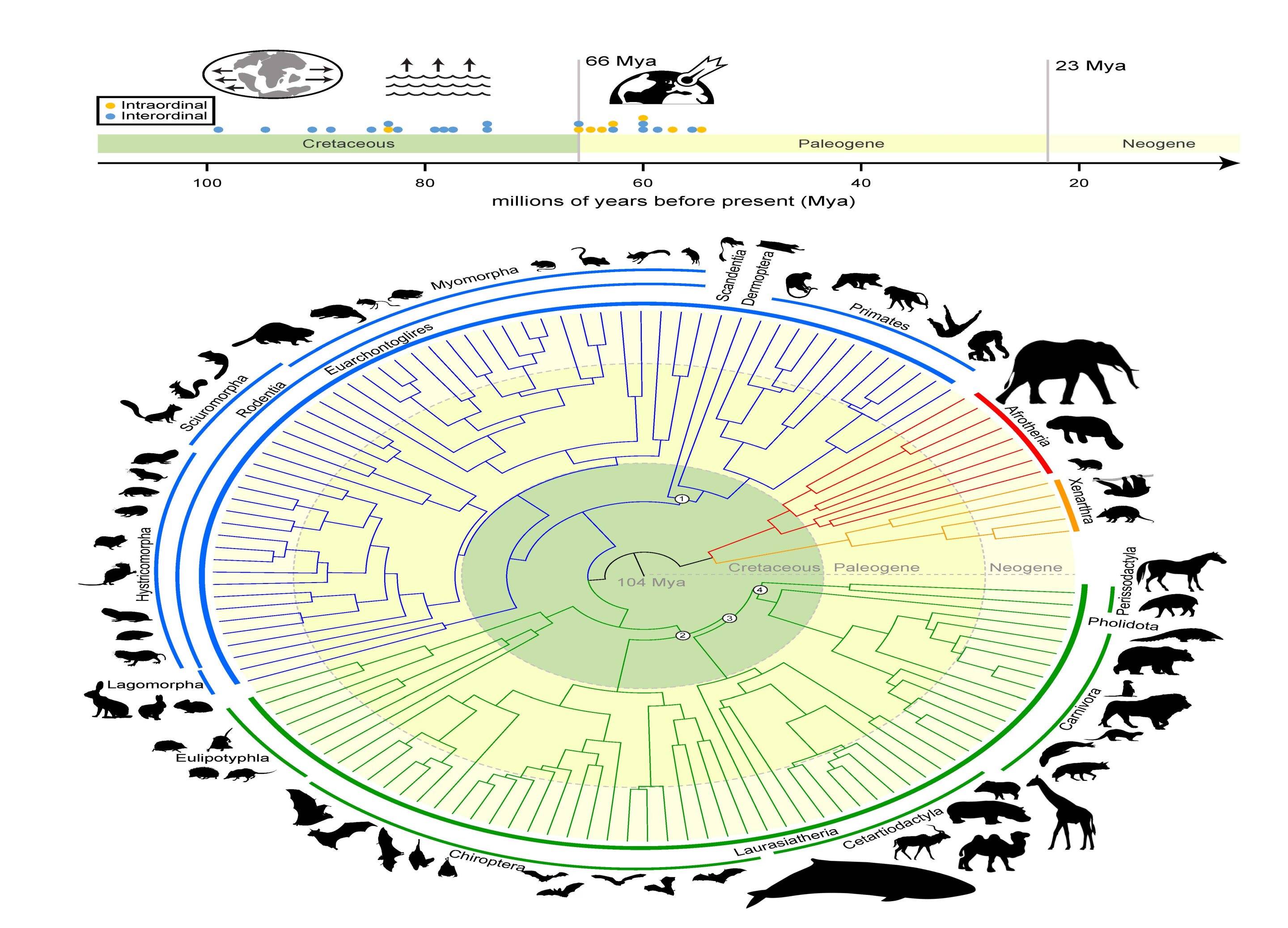
„Das zentrale Argument dreht sich darum, ob Plazenta-Säugetiere (Säugetiere, die sich in einer Plazenta entwickeln) vor oder nach dem Aussterbeereignis der Kreidezeit und des Paläogens (oder K-Pg) auseinandergingen, das die Nicht-Vogel-Dinosaurier auslöschte“, sagte Foley Aufgrund des enormen Ausmaßes von Zoonomia beantworten wir die Frage, wo und wann sich Säugetiere in Bezug auf das Massensterben von K-Pg diversifiziert und entwickelt haben.
Die Forschung – die mit Mitarbeitern an der University of California, Davis durchgeführt wurde; Universität von Kalifornien, Riverside; und das American Museum of Natural History – kommt zu dem Schluss, dass die Diversifizierung der Säugetiere vor dem K-Pg-Aussterben als Folge der Kontinentaldrift begann, die dazu führte, dass Landmassen auf der Erde drifteten und über Millionen von Jahren wieder zusammenkamen. Ein weiterer Diversifizierungsschub ereignete sich direkt nach dem Aussterben der K-Pg-Dinosaurier, als Säugetiere mehr Platz, Ressourcen und Stabilität hatten.
Diese beschleunigte Diversifizierungsrate hat zu der reichen Vielfalt von Säugetierlinien geführt – wie Fleischfresser, Primaten und Huftiere – die heute die Erde teilen.
Die Forschung von Murphy und Foley wurde von der National Science Foundation finanziert und ist Teil des Zoonomia-Projekts unter der Leitung von Elinor Carlson und Kirsten Lindblad-Toh vom Broad Institute, das auch Säugetiergenome vergleicht, um die Grundlage bemerkenswerter Phänotypen zu verstehen – die Expression bestimmter Gene wie braune Augen versus blaue Augen – und die Ursprünge von Krankheiten.
Foley bemerkte, dass die Vielfalt unter Plazentasäugern sowohl in ihren körperlichen Merkmalen als auch in ihren ungewöhnlichen Fähigkeiten offensichtlich ist.
„Säugetiere stellen heute eine enorme evolutionäre Vielfalt dar – vom brutzelnden Flug der winzigen Bienenfledermaus bis zum trägen Gleiten des riesigen Blauwals, der durch die riesigen Ozeane der Erde schwimmt. Mehrere Arten haben sich zu Echoortungen entwickelt, von denen einige Gift produzieren, während andere sich entwickelt haben Krebs Virale Resistenz und Toleranz „.
„In der Lage zu sein, die Unterschiede und Ähnlichkeiten von Säugetierarten auf genetischer Ebene zu betrachten, kann uns dabei helfen, herauszufinden, welche Teile des Genoms notwendig sind, um die Expression von Genen zu regulieren“, fügte sie hinzu. „Die Modifikation dieser genomischen Maschinerie bei verschiedenen Arten hat zu der Vielfalt von Merkmalen geführt, die wir heute bei lebenden Säugetieren sehen.“
Murphy teilte mit, dass die aufgelöste Foley-Linie von Säugetieren entscheidend für die Ziele des Zoonomia-Projekts ist, das darauf abzielt, die Leistungsfähigkeit der vergleichenden Genomik als Werkzeug für die Humanmedizin und den Erhalt der biologischen Vielfalt zu nutzen.
„Das Zoonomia-Projekt ist wirklich wirkungsvoll, weil es die erste Analyse ist, die gleichzeitig 241 verschiedene Säugetiergenome angleicht und diese Informationen verwendet, um das menschliche Genom besser zu verstehen“, erklärte er. „Die Hauptmotivation für die Zusammenstellung dieses riesigen Datensatzes bestand darin, all diese Genome mit dem menschlichen Genom vergleichen zu können und dann die Teile des menschlichen Genoms zu identifizieren, die sich im Laufe der Evolutionsgeschichte der Säugetiere verändert haben.“
Die Identifizierung, welche Teile von Genen manipuliert werden können und welche Teile nicht verändert werden können, ohne die Genfunktion zu beeinträchtigen, ist wichtig für die Humanmedizin. Eine aktuelle Studie in Translationale Medizinwissenschaften Unter der Leitung von einem Kollegen von Murphy und Foley, dem Genetiker Dr. Scott Dindot von Texas A&M, verwendete er einen vergleichenden genomischen Ansatz zur Entwicklung Molekulare Therapie des Angelman-SyndromsEs ist eine seltene und verheerende angeborene neurologische Störung, die durch den Verlust der mütterlichen Funktion verursacht wird UBE3A Gen im Gehirn.
Dindots Team nutzte die gleichen Maße für evolutionäre Einschränkungen, die vom Zoonomia-Projekt identifiziert wurden, und wendete sie an, um ein wichtiges, aber zuvor unbekanntes genetisches Ziel zu identifizieren, das zur Rettung der Expression von verwendet werden könnte UBE3A in menschlichen Nervenzellen.
Murphy sagte, dass die Erweiterung der Fähigkeit zum Vergleich von Säugetiergenomen unter Verwendung des größten Datensatzes in der Geschichte dazu beitragen wird, mehr Behandlungen und Therapien für Krankheiten anderer Arten zu entwickeln, die in der Genetik verwurzelt sind, einschließlich Katzen und Hunden.
„Zum Beispiel haben Katzen physiologische Anpassungen, die in einzigartigen Mutationen verwurzelt sind, die es ihnen ermöglichen, eine fettreiche, proteinreiche und für Menschen äußerst ungesunde Ernährung zu sich zu nehmen“, erklärte Murphy. „Einer der netten Aspekte von Zoonomias Ausrichtung von 241 Arten ist, dass wir jede Art (nicht nur Menschen) als Referenz auswählen und bestimmen können, welche Teile des Genoms dieser Art sich frei verändern können und welche Veränderungen nicht tolerieren Bei Katzen könnten wir beispielsweise helfen, genetische Anpassungen bei diesen Arten zu identifizieren, die zu therapeutischen Zielen für Herz-Kreislauf-Erkrankungen beim Menschen führen könnten.
Murphy und Foley waren auch maßgeblich an vielen der späteren Arbeiten beteiligt, die Teil des Projekts sind.
„Es ist gestaffelte Genomik“, erklärte Foley. „Eines der befriedigendsten Dinge für mich, als Teil eines breiteren Projekts zu arbeiten, ist zu sehen, wie viele verschiedene Forschungsprojekte durch die Einbeziehung unserer Version in ihre Analysen ergänzt wurden. Dies schließt Studien zur Erhaltungsgenomik gefährdeter Arten ein die Evolution verschiedener komplexer menschlicher Eigenschaften. „.
Foley sagte, es sei lohnend und lohnend, die heiß diskutierte Frage nach dem Zeitpunkt des Ursprungs von Säugetieren und der Produktion einer erweiterten Abstammungslinie, die die Grundlage für die nächsten Generationen von Forschern legt, endgültig zu beantworten.
„In Zukunft wird dieser massive Genomabgleich und seine historische Aufzeichnung der Evolution des Säugetiergenoms die Grundlage für alles sein, was jeder tun wird, wenn er vergleichende Fragen zu Säugetieren stellt“, sagte sie. „das ist toll“.
Mehr Informationen:
Nicole M. Foley et al., Eine genomische Zeitskala der Plazenta-Säugetierentwicklung, Wissenschaften (2023). DOI: 10.1126/science.abl8189
Zeitschrifteninformationen:
Translationale Medizinwissenschaften

„Böser Kaffee-Nerd. Analyst. Unheilbarer Speckpraktiker. Totaler Twitter-Fan. Typischer Essensliebhaber.“
